Les espèces sont toutes apparentées. cependant, on considère que les espèces les plus apparentées sont celles qui présentent le plus grand nombre de caractères homologues dérivés (ou évolués) communs : elles l’ont hérité du dernier ancêtre qu’elles ont en commun. Un caractère dérivé provient d’une innovation évolutive qui a affecté le caractère à l’état primitif (ou ancestral).
I- Phylogénie à partir de données anatomiques
La représentation de ces liens de parenté se fait par des arbres phylogénétiques :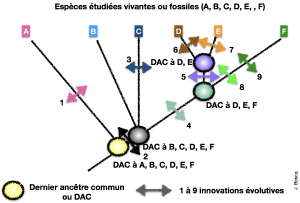
★chaque branche est terminée par une espèce fossile ou vivante (A à F)
★sur certaines branches, sont indiquées les innovations évolutives : apparition, disparition, transformation d’un caractère (1à6)
★Les nœuds (points de séparation entre plusieurs branches) représentent les ancêtres communs hypothétiques : ce ne sont pas des fossiles mais leur existence est avérée par simple raisonnement logique.
1- Utiliser les données anatomiques fournies par le logiciel Phylogène dans la collection « Archontes » afin de préciser les relations de parenté entre le Chimpanzé, le Gibbon, le Gorille, l’Homme, le Maki, la Macaque, l’Ourang-Outan, le Saki et le Tarsien.
Vous considèrerez le Toupaïe : il n’appartient pas au groupe des primates, il possède tous les caractères à l’état primitif. Vous considèrerez tous les caractères sauf le caractère « knucckle walking ».
Pour cela, veuillez suivre les étapes suivantes en utilisant les fonctionnalités du logiciel Phylogène (voir fiche technique) :
a) Construire une matrice de caractères : il s’agit de réaliser un tableau de comparaison des différents attributs de chaque espèce.
b) Polariser et coder les états des caractères : cela revient à mettre en évidence dans le tableau les attributs qui sont des caractères primitifs d’une couleur et ceux qui sont des caractères dérivés d’une autre couleur.
c) Construire un arbre phylogénétique.
 Appeler le professeur pour vérification !
Appeler le professeur pour vérification !
Après vérification, allez chercher la feuille « Etude des données anatomiques – Document obtenu grâce à l’utilisation du logiciel Phylogène » au bureau : Annexe phylogène
2- Indiquer sur l’arbre phylogénétique les innovations évolutives.
3- a) Sur l’arbre phylogénétique, mettre en évidence le dernier ancêtre commun aux primates.
b) Donner ses caractéristiques.
4- a) Quels sont les primates avec qui l’Homme est le plus proche parent ?
b) Comment pourrait–on préciser les liens de parenté entre ces êtres vivants ?
2- Indiquer sur l’arbre phylogénétique les innovations évolutives.
3- a) Sur l’arbre phylogénétique, mettre en évidence le dernier ancêtre commun aux primates.
b) Donner ses caractéristiques.
4- a) Quels sont les primates avec qui l’Homme est le plus proche parent ?
b) Comment pourrait–on préciser les liens de parenté entre ces êtres vivants ?
 Appeler le professeur pour vérification !
Appeler le professeur pour vérification !
II – Phylogénie à partir de données moléculaires
1- Utiliser les données moléculaires (séquences de la protéine COX2, protéine impliquée dans la respiration cellulaire) fournies par le logiciel Phylogène afin de préciser les relations de parenté entre les grands primates c’est–à–dire l’Homme et ses plus proches parents identifiés lors de l’étude A-.
Pour cela, suivre les étapes suivantes en utilisant les fonctionnalités du logiciel Phylogène (voir la fiche technique) :
a) Afficher une matrice de distances : il s’agit d’obtenir un tableau des différences (ici du nombre de différence) entre les séquences de la molécule considérée entre les espèces considérées
b) Afficher un arbre : il s’agit d’un arbre phylogénétique que le logiciel construit à l’aide des données de la matrice des distances
 Appeler le professeur pour vérification !
Appeler le professeur pour vérification !
Après vérification, allez chercher la feuille « Etude des données moléculaires – Document obtenus grâce à l’utilisation du logiciel Phylogène » au bureau. Annexe phylogène
2- Concernant l’étude portant sur la protéine COX2 : Montrer que l’arbre est en accord avec les données de la matrice des distances.
Plus la similitude entre les séquences de molécules est importante, plus les espèces qui possèdent ces molécules sont proches.
3- a) Comparer les relations de parenté établies grâce à la comparaison des séquences de la protéine COX2 et celle de la protéine protamine, protéine se trouvant dans le noyau de spermatozoïdes.
b) Comment trancher ?
Remarque importante : Voici ce qu’a écrit Philippe Janvier du laboratoire de paléontologie du Muséum d’Histoire Naturelle de Paris :
Ainsi, à partir des années 1980, l’utilisation des séquences moléculaires de gènes dans la reconstruction de phylogénies a pu être considérée comme un test des arbres construits sur la base de la seule distribution des caractères morphologiques. Mais la confrontation de seulement deux sources de données ne peut guère emporter une décision. Il ne peut
y avoir que contradiction ou concordance. On considère pourtant que la concordance entre une phylogénie morphologique et une phylogénie moléculaire équivaut à une corroboration de l’hypothèse de relations de parenté. Est-ce réellement le cas? n’est-ce pas un “match nul”? Une troisième source de données serait nécessaire pour faire le départ. Peut-être cette nouvelle source de données sera-t-elle issue de la génétique du développement, héritière de l’embryologie qui, depuis von Baer, Serre et Agassiz, au XIXe siècle, a souvent été l’arbitre du degré de généralité des caractères.
Une seule chose est certaine, dans les limites de la science actuelle : nous ne pouvons repasser le “film” de l’évolution, tout comme nous ne pouvons repasser celui de l’histoire. La transformation d’une nageoire en membre est un événement tout aussi inaccessible à l’observation directe qu’un évènement historique assez ancien pour n’avoir plus de témoins oculaires. Nous n’avons que des documents, des caractères, qu’ils soient “historiques”, morphologiques, ou moléculaires. Nous choisissons donc de leur faire confiance, car ils sont globalement cohérents dans leur répartition hiérarchique, donc plus satisfaisants que la mythologie.
Philippe Janvier – UMR 8569, Laboratoire de Paléontologie, Muséum National d’Histoire Naturelle, Paris d’après CNRS
